Secuenciación de alto rendimiento como herramienta para monitorizar comunidades de procariotas en una planta de tratamiento de aguas residuales
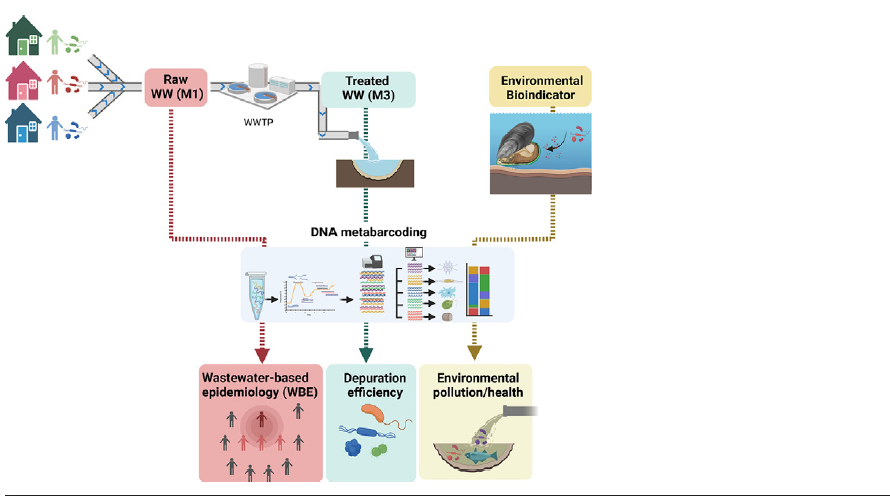
En este estudio, se utilizó la técnica de metabarcodificación de ADN para explorar la diversidad de procariotas y la estructura de la comunidad en aguas residuales recolectadas en primavera e invierno 2020–2021, así como la eficiencia del tratamiento en un sistema de aguas residuales depuradora (EDAR) en la Ría de Vigo (NO de España). Los muestreos incluyeron aguas residuales sin tratar de la corriente de entrada (M1), el agua de vertido tras el tratamiento de desinfección (M3) y los mejillones utilizados como bioindicadores de posible contaminación del medio marino. Se descubrieron diferencias significativas en el microbioma de cada tipo de muestra (M1, M3 y mejillones), con un 92 %, 45 % y 44 % de OTU exclusivas encontradas en muestras de mejillón, M3 y M1 respectivamente.
También se detectaron diferencias estacionales en las muestras de aguas residuales, con lo cual los parámetros (temperatura, pH) podría estar fuertemente involucrados. Las bacterias presentes en las aguas residuales sin tratar (M1) se asociaron con el microbioma intestinal humano y por tanto, se detectaron potenciales patógenos que podrían estar circulando en la población en periodos específicos (por ejemplo, Arcobacter sp. y Clostridium sp.). Una disminución considerable en los organismos patógenos putativos de M1 a
Participantes
Universidad de Vigo, Instituto de Investigacións Mariñas CSIC , GESECO AGUAS S.A.
Fecha: Mayo 2020 – DICIEMBRE 2022